草原植物25種のトランスクリプトーム全長解析
以下は、小生が知らなかったのだが、単なる25種の草原の植物の全遺伝子を読み込んだデータを作ったという論文である。これまで遺伝子を読んだだけのデータは論文になりにくかったのだが、それが掲載可能な雑誌「Scientific Data 」なるものが存在することを始めて知った。以下はドイツのグループのデータである。必ず役に立つときが来ると思う。
紙面の都合上、結果の、表1,2,3のみ記した。
草原植物25種のトランスクリプトーム全長解析
Full-length transcriptomes of 25 grassland plant species
Chongyi Jiang 1 ,Zixia Huang 2, Cynthia Meizoso3, Gaby Kumpfmüller3, Jochen B. W. Wolf3,4 & Holger Schielzeth 1,4
1Population ecology Group, institute of ecology and evolution, friedrich Schiller University, Jena, Germany. 2School of Biology and environmental Science, University college Dublin, Dublin, ireland. 3Division of evolutionary Biology, Faculty of Biology, LMU Munich, Grosshaderner Str. 2, 82152, Planegg-Martinsried, Germany.
Scientific Data volume 12, Article number: 922 (2025)
草原は、重要な生態系サービスを提供する不可欠な生物多様性生態系である。その生態学的・経済的価値にもかかわらず、生態進化学的・機能ゲノム学的研究をサポートする野生草原種のトランスクリプトームリソースは依然として限られている。ここでは、長期にわたる生物多様性実験(イエナ実験)から収集した25種の野生草原植物のシュート組織の全長トランスクリプトームを紹介する。PacBio Iso-Seq技術を用いて、合計5億2,245万個のサブスレッドを作成した。これらのサブスレッドは、種ごとに独立してユニークな転写産物にアセンブルされた。その結果、1種あたり平均49,180の転写産物が得られ、そのうち68.6%はSwiss-Protデータベースを用いて、アノテーションに成功した。さらに、転写物の40.3%は完全なオープンリーディングフレーム(ORF)を含んでいたが、31.4%は不完全なORFであった。36.8%以上の転写産物が非コードRNAとして同定された。全トランスクリプトームにおいて、平均5.08%の塩基が繰り返し要素と判定された。このデータセットは、草原種における遺伝子発現、代替スプライシング、進化パターンを研究するための貴重な全長トランスクリプトームリソースを提供し、機能ゲノミクスと保全における将来の研究への道を開くものである。
背景と概要
草原生態系は、地球上で最も生物多様性の高いバイオームのひとつであり、炭素隔離、土壌肥沃度、野生生物の生息に重要な役割を果たしている。しかし、草原生態系は、気候変動、土地の改変、生息地の分断化によってますます脅かされている。草原植物の機能ゲノム研究により、これらの生態系が環境変化にどのように対応するかをより深く理解し、効果的な保全戦略の開発を支援することができる。
次世代シーケンサー(NGS)技術は、迅速かつ費用対効果の高いゲノムおよびトランスクリプトーム解析を可能にし、生物学研究に革命をもたらした。その中でも、Pacific Biosciences社が開発した1分子リアルタイム(SMRT)シーケンスに基づくロングリードアプローチであるIsoform sequencing(Iso-Seq)は、全長転写産物の捕捉を可能にし、アイソフォーム、代替スプライシングイベント、完全な遺伝子モデルの詳細な特性解析を容易にしている。これらの技術は植物研究、特に経済的に重要な食用作物において広く応用され、家畜化や病害抵抗性の遺伝的基盤に関する理解を大きく前進させてきた。しかし、非モデル種、特に野生の草原植物に関するゲノムおよびトランスクリプトーム研究は依然として限られている。草原生態系における遺伝子の機能、適応、回復力についての理解を深めるためには、これらの種の包括的なトランスクリプトームデータが決定的に必要である。
ここでは、PacBio Iso-Seqテクノロジーを用いて作成した、系統学的網羅範囲の広い25種の野生草原植物のトランスクリプトームインベントリーを紹介する(表1)。これらの25種は、中央ヨーロッパの草原、特に長期のイエナ(Jena)実験において支配的な分類群であり、広範な生態学的データが蓄積されている。合計5億2,245万個のサブスレッドを作成し、それらをアセンブルした後、Swiss-Protデータベースを用いてアノテーションを行った。さらに、非コードRNA(ncRNA)、コード配列、トランスポーザブルエレメントを全種において同定した。本データセットは、野生の草原生物種における初の大規模な全長転写産物コレクションであり、これらの生態学的に重要な生物における遺伝子構造、アイソフォームの多様性、進化的関係性についての貴重な知見を提供するものである。
方法
サンプル収集とRNA調製。
植物サンプルは、長期草原生物多様性フィールドサイトであるイエナ実験38から収集した。この研究では、RNA抽出と配列決定のために、選ばれた25種の草原からシュート組織を収穫した
(表1)。各サンプルから約10 mgの組織を、
-80℃の低温QIAGEN Tissuelyser IIでホモジナイズした。Lathyrus pratensis、Trifolium
pratense、Daucus carota、Primula veris、Luzula campestris、Lotus corniculatusの全RNAは、TRIzol試薬(Zymo Research、Direct-zol RNA Microprep Kits #R2062)を用いて、製造元のプロトコールに従って抽出した。残りの種については、β-メルカプトエタノールを含む溶解バッファー(Roboklon, Universal RNA Purification Kit #E3598)を用いて RNA を抽出した。すべてのサンプルについて、OneStep PCR Inhibitor Removal Kit(Zymo Research)を用いてクリーンアップステップを行った。RNA の純度と濃度は NanoDrop ND-1000 spectrophotometer と QubitTM (Thermo Fisher Scientific) を用いて評価した。RNAの完全性は、サンプルを1.2%アガロース上で測定することで確認した。
PacBioライブラリーの構築とシーケンス。
PacBio Iso-Seqライブラリーは、Pacific Biosciences社が提供する
標準Iso-Seqライブラリー調製プロトコールに従って構築した。SMARTer® PCR cDNA Synthesis Kit(タカラバイオ)を用いて全長cDNAを合成し、PCR増幅と精製を行った。合成されたcDNAは、損傷修復と末端修復が行われた。SMRTbellアダプターを二本鎖cDNA分子の末端にライゲーションした。Sequel Binding and Internal Control Kit 3.0(Pacific Biosciences)を用いて、プライマーとポリメラーゼをライブラリーに結合させ、塩基配列を決定した。最終ライブラリーの精製はAMPure PB magnetic beads(Pacific Biosciences社製)を用いて行い、ライブラリーをPacBio Sequel IIシステムにロードして塩基配列を決定した。生シーケンスデータは、
13.64Gbpから31.67Gbpの範囲であり、15,119,410から26,635,333サブスレッドに相当した(表2)。リード収量のばらつきは主にRNA抽出効率の違いによるもので、トランスクリプトームの完全性に影響を与えた可能性がある。
Pacbio Iso-seqデータ処理。
Circular Consensus Sequences (CCS)は、ccs tool (version 6.4.0)を使用し、デフォルトのパラメータでサブリードから生成した。このルーチンは、各分子の複数回のパスに基づいて、高忠実度の全長リードを同定する。1種あたり平均191,105のCCSリードが得られた(表2)。これらのCCSリードをさらにlima(バージョン2.9.0、-isoseqオプションを使用)で処理し、シーケンシングアダプターとバーコードを除去した。isoseqツール(バージョン4.0.0)を用いてPoly(A)テールと人工コンカテマーを除去し、Full-Length Non-Chimeric(FLNC)リードを得た。このFLNCリードをisoseq clusterを用いてクラスタリングし、Pacbio Iso-seqデータ処理に用いた。PacBio Iso-seqパイプラインを用いて生シーケンスデータを処理した。ccs tool (version 6.4.0)を用い、デフォルトのパラメータでサブスレッドからCircular Consensus Sequences (CCS)を生成した。このルーチンは、各分子の複数回のパスに基づいて、高忠実度の全長リードを同定する。1種あたり平均191,105のCCSリードが得られた(表2)。これらのCCSリードをさらにlima(バージョン2.9.0、-isoseqオプションを使用)で処理し、シーケンシングアダプターとバーコードを除去した。isoseqツール(バージョン4.0.0)を用いてPoly(A)テールと人工コンカテマーを除去し、Full-Length Non-Chimeric(FLNC)リードを得た。このFLNCリードをisoseq clusterを用いてクラスタリングし、シングルトン転写産物を保持するsingletonsオプションを有効にして、ポリッシュ転写産物を生成した。1種あたり平均49,180 FLNCリードが生成され(表2)、リード長は51〜8,685 bpであった(図1)。
機能アノテーション。
全長転写産物のアノテーションを行うために、BLASTx(バージョン2.14.0)39を用いて、FLNCリードをSwiss-Protタンパク質データベース(リリース2024-07-24)に対してアライメントした。BLASTx検索はE値カットオフ1×10-10で行われ、相同タンパク質を正確に同定することができた。この
アノテーションにより、トランスクリプトームで検出された転写産物の機能的洞察が得られた。種あたりの転写産物の平均68.6%がアノテーションに成功し、これは平均9,128個のユニークなタンパク質に対応した(各生物種の表2を参照)。マルチプレット転写産物(少なくとも2つのFLNCリードでサポートされるもの)は、シングルトン転写産物(1つのFLNCリードでサポートされるもの)と比較して、一貫して高いアノテーション率を示したが、その差は10%未満であった(図1)。BUSCO(Benchmarking Universal Single-Copy Orthologs)40を用いて、アセンブルした転写産物の完全性をさらに評価した。比較のために、シロイヌナズナの葉の転写産物データも含めた。リード(GEO accession: GSM6589889)41は、Salmon(v1.10.2)を用いてシロイヌナズナのコード配列(CDS)にマッピングした。CDSリファレンスはTAIRデータベース(Araport11 CDS, 2022-0914)42から取得した。この比較分析により、草原
植物におけるトランスクリプトームの質と完全性を、よく特性化されたモデル種と比較して評価することができる(図1)。
オープンリーディングフレーム(ORF)とノンコーディングRNA(ncRNA)の予測。
ORF予測はTransDecoder(v5.7.1)を用いて行った。その結果、開始コドンと停止コドンの両方を含む完全なORFが40.3%、部分的なORFが31.4%同定された(図2)。非コードRNA(ncRNA)は、CPC2(v 0.1)43とPINC44を用いて予測した。両ツールによって予測されたncRNAは、有意な重複を示した。平均して、2つのツールによって予測されたオーバーラップは、転写産物の36.8%がノンコーディングRNAであることを示している(図2)。TransDecoderがORFを同定しなかった転写産物のうち、96.7%(ranging from 95% to 98%)がCPC2とPINCによってもncRNAと予測された。
反復エレメントの予測。
RepBase46(20181026リリース)とDfam47(3.1リリース)を組み合わせたViridiplantaeライブラリーを用いて、RepeatMasker45(バージョン4.1.7-p1)を用いてトランスクリプトーム内の繰り返し要素を同定した(表3)。25種の平均で、塩基の5.08%が反復要素としてフラグ付けされた。このうち1.96%はレトロエレメントとして分類され、1.03%は低分子RNAとして分類された。反復エレメントの分布をさらに調べるため、反復エレメントと同定された塩基の割合に基づいて転写産物を分類した(0-0.1%、0.1-10%、10-50%、50-100%)。この解析は、25の生物種にわたって、コード配列と非コード配列について別々に行った(表3)。
表1. 対象種。
表2. トランスクリプトーム統計のまとめ。
表3. 様々な割合の反復要素を含む転写産物の割合。
転写産物は、繰り返し要素と判定された塩基の割合(0-0.1%、0.1-10%、10-50%、50-100%)に基づいて4つのグループに分類されている。*ノンコーディングRNAはCAPTとCPC2によって同定されたオーバーラップであり、**コーディングRNAはそれ以外の転写産物である。数字は塩基の割合。
参考までに(森敏記述):Jena Experiment プロジェクトについて。
イエナ実験は、2002年に設立された長期草地生物多様性実験(DFG FOR456/1451)であり、短期的および長期的な生物多様性と生態系機能の関係の基礎となるメカニズムを理解することを主な目的としている。イエナ実験の枠組みにおいて、私たちは植物群落の発展や、植物群落の多様性に対する個々の植物種の反応の研究を行っている。最近では、植物群落の多様性、植物の歴史、土壌の歴史に対する植物種の反応について、進化過程の役割を研究している。現在の研究の焦点は、植物の多様性が植物の栄養応答の時間的動態に及ぼす影響に関する研究であり、その安定性には、時間的長期不変性、環境摂動時の抵抗性、環境摂動後の回復性などが含まれる。
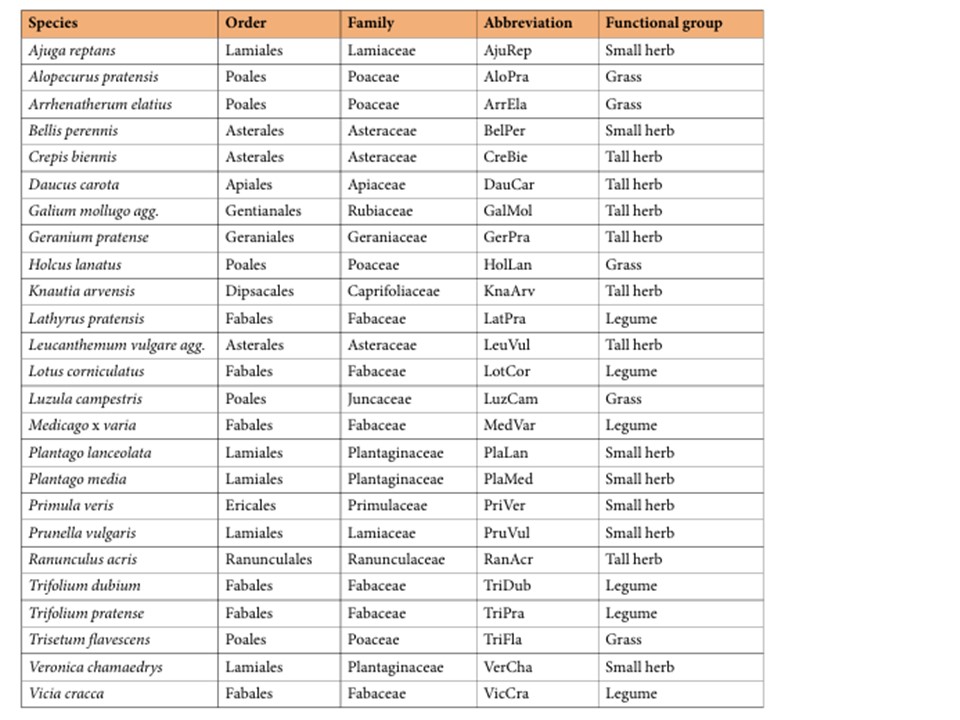
表1
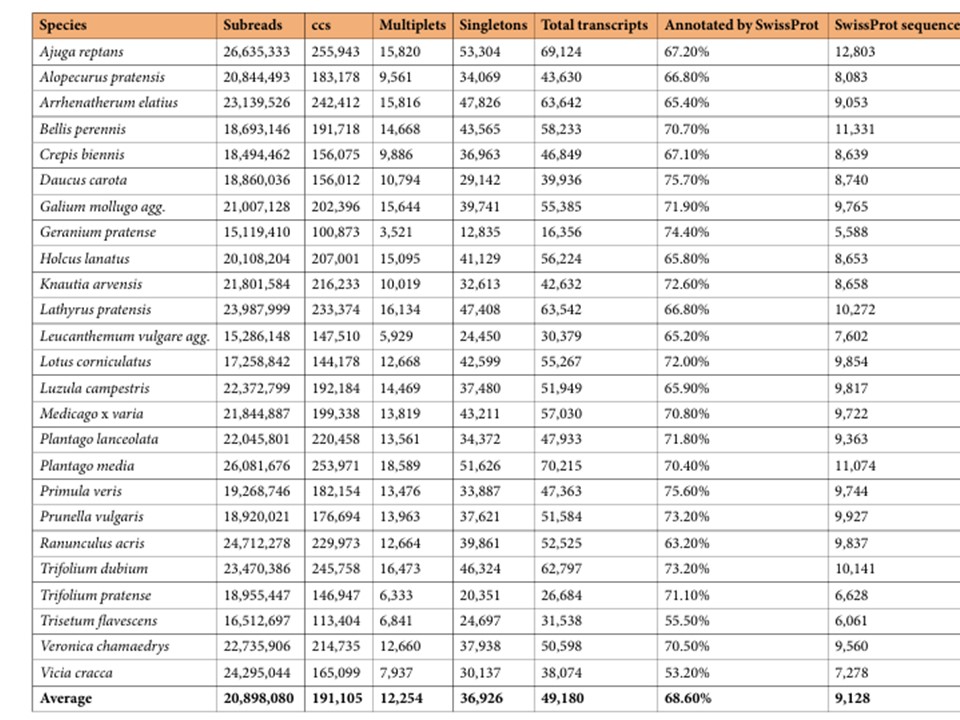
表2
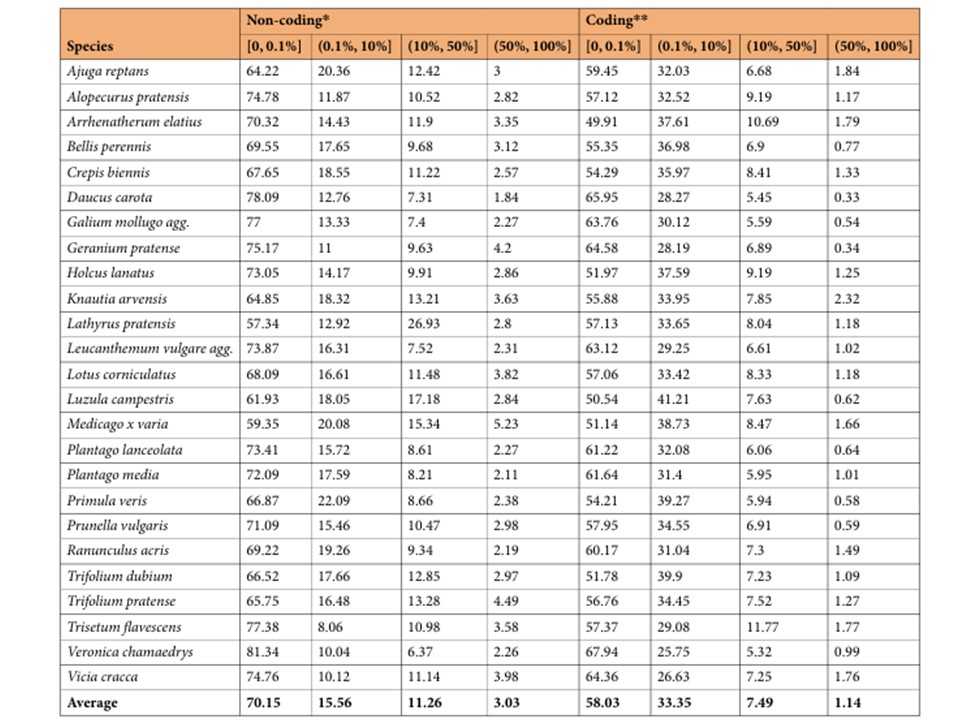
表3