鉄欠乏応答制御因子IDEF2における保存された翻訳抑制的上流オープンリーディングフレームの発見
以下の論文に出てくるIDEF2は小郷さん(Ogo)が見つけたものであるが、この遺伝子自身の制御機能が解明されていなかった。したがって以下の論文は重要と考えて、要約、まえがき、論議の3か所を全訳しておいた。紙面の都合で図3と図4は紹介できていない。
鉄欠乏応答制御因子IDEF2における保存された翻訳抑制的上流オープンリーディングフレームの発見
Discovery of a conserved translationally repressive upstream open reading frame within the iron-deficiency response regulator IDEF2
Oscar Carey-Fung, Jesse T. Beasley, Ronan C. Broad, Roger P. Hellens and Alexander A. T. Johnson
BMC Plant Biology (2024) 24:891 https://doi.org/10.1186/s12870-024-05473-y
--------
要旨
背景 : 鉄(Fe)欠乏症は、世界人口の30〜50%に影響を及ぼしている。主食作物の遺伝的バイオフォーティフィケーションは、人間の栄養状態を改善するための有望な戦略であるが、鉄のバイオフォーティフィケーションに有効な精密育種ターゲットの数は少ない。アップストリームオープンリーディングフレーム(uORF)は、遺伝子の5'リーダー配列(5’LS)内のシス制御要素であり、一般にメインオープンリーディングフレーム(mORF)の翻訳を抑制する。
結果 : イネ(Oryza sativa L.)のribo-seqデータセットとトランスクリプトームをアライメントし、重要なFeホメオスタシス遺伝子内の推定uORFを同定した。デュアルルシフェラーゼアッセイ(DLA)を用いて、これらのuORFがmORFの翻訳抑制を引き起こすかどうかを決定し、mORFの翻訳抑制を解除するために変異させることができるLS領域を特定した。
翻訳抑制性uORF領域は、鉄欠乏応答の2つのポジティブレギュレーターで同定された: IDEF1とIDEF2である。IDEF2-uORFペプチドは単子葉植物間で高度に保存されており、コムギ(Triticum aestivum L.)TaIDEF2-A1遺伝子の5'LSにおける一連の変異は、可変的なmORF抑制を示した。
結論 :これらの結果は、IDEF2転写因子が単子葉植物における鉄欠乏応答を調節する可能性のある調節機構を明らかにし、作物の栄養および生物的ストレス耐性を改善するための新たな精密育種ターゲットを浮き彫りにした。
ハイライト:作物栄養改善のための精密育種ターゲットを見つけるために、鉄ホメオスタシス遺伝子内の翻訳抑制性uORFを探索し、IDEF1とIDEF2にuORFを発見した。 ------------
キーワード バイオフォーティフィケーション、CPuORF、デュアルルシフェラーゼ、IDEF1、イネ、転写因子、uORF、コムギ
はじめに
植物の鉄ホメオスタシスは、鉄キレーター、トランスポーター、貯蔵タンパク質の発現を制御する転写因子のネットワークによって維持されている。ニコチアナミンは、すべての高等植物において長距離鉄輸送を促進する鉄キレーターであり、イネ科単子葉植物では分泌型植物シデロフォアである2'-デオキシムギニン酸(DMA)の前駆体として機能する。根圏では、鉄-DMA複合体は黄色ストライプ様(YSL)輸送体によって根に吸収される。ニコチアナミン合成酵素(NAS)またはDMA合成酵素(DMAS)遺伝子のノックアウトまたはノックダウンは、Fe欠乏に対する感受性を低下させ、植物成長を低下させるが、イネOsNAS2遺伝子の過剰発現は、農学的パフォーマンスに影響を与えることなく、イネおよびパンコムギ(Triticum aestivum L.)の穀物Fe濃度を増加させる。鉄ホメオスタシス遺伝子の発現は、鉄関連転写因子2および3(IRO2、IRO3)、鉄ホメオスタシスのポジティブレギュレーター1、2および3(PRI1/2/3)など、いくつかの塩基性ヘリックス-ループ-ヘリックス(bHLH)転写因子によって制御されている。一方、OsIRO2 または OsPRI2 遺伝子を過剰発現させると、鉄欠乏応答がアップレギュレートされ、 ストレス耐性が向上し、穀物中の鉄濃度が野生型(WT)植物に対して 1.5 倍から 2 倍に増加する。対照的に、ネガティブレギュレーターであるOsIRO3のノックアウトまたは過剰発現は、いずれも鉄欠乏に対して過敏となる。鉄欠乏応答の他の正の制御因子にはIRONMAN(IMA)ファミリーがあり、イネで人工IMA遺伝子を過剰発現させると穀物中の鉄濃度が2倍に上昇する一方、シロイヌナズナでIMA遺伝子ファミリーをサイレンシングすると鉄の取り込みが阻害される。ヘメリトリン(haemerythrin)モチーフを含む実に興味深い新遺伝子(RING)およびジンクフィンガータンパク質(HRZ1およびHRZ2)は、鉄レベルを感知し、bHLH転写因子を分解および鉄欠乏応答の減衰の標的とし、イネにおけるOsHRZ2のノックダウンは、穀物鉄濃度を3.8倍まで増加させた。単子葉植物における鉄欠乏応答の2つの正の制御因子は、ABI3/VP1転写因子ファミリーのメンバーである鉄欠乏応答性エレメント(IDE)結合因子1(IDEF1)と、NAC転写因子ファミリーのメンバーであるIDE結合因子2(IDEF2)である。IDEF1およびIDEF2転写因子は、いくつかの鉄ホメオスタシス遺伝子のプロモーター領域のIDEに結合し、それらの発現をアップレギュレートする。OsIDEF1とOsIDEF2の両方の発現はイネにおいて構成的であり、異なる環境鉄条件下で保存されており、OsIDEF1は鉄イオンと直接結合することができる。対照的に、IDEF2がIDE結合のために調節されるメカニズムは不明である。遺伝子の5'リーダー配列(LS)から始まるアップストリームオープンリーディングフレーム(uORF)は、メインORF(mORF)を転写後に制御するシス制御要素である。植物種におけるuORFを含む転写産物の割合は6〜48%であり、メインORFの翻訳開始部位(mAUG)の上流にAUG配列(uAUG)が存在するか、リボソームプロファイリングによって上流翻訳が検出されることで予測される。保存ペプチドuORF(CPuORF)は、代謝機能を持つペプチドを産生する可能性が高いuORFであり、文献の中にはよく特徴付けられたCPuORFが多数あるにもかかわらず、非保存ペプチドuORFがすべてのuORFの大部分を形成している。一部のuORFはmORFの翻訳を増加させることが示されているが、ほとんどのuORFはノイズを減らし、エネルギーを節約する手段として、翻訳を減衰させる働きをする。uORF を変異させると、一般に mORF の翻訳効率が向上するため、遺伝子発現操作や作物育種のための貴重な遺伝子編集ターゲットとなるが、uORF 同定は依然として大きなハードルとなっている。植物のuORFを同定する主な方法には、相同性ベースのアプローチ(uORFinder、uORFSCAN、BAIUCAS/ESUCA)、リボソームプロファイリング(psORF)、ペプチドゲノミクス(psORF)などがある。まれな例では、GWASプロテオミクスアプローチにより、トウモロコシ(Zea mays L.)内のタンパク質存在量を変化させるいくつかの天然のuORFバリアントが同定された。リボソームプロファイリングは、翻訳イベントの偏りのないスナップショットを提供するために、リボソームで保護されたフラグメントのディープシーケンスを使用し、非正規ORF部位で暗号化された翻訳イベントを検出することができる。本研究では、精密育種ターゲットとして単子葉植物のFeホメオスタシス遺伝子内の翻訳抑制性uORFの探索と、IDEF1とIDEF2におけるuORFの発見について述べる。IDEF1-uORFはイネにのみ存在し、環境鉄の変化に反応しないのに対し、IDEF2-uORFペプチドはほとんどの単子葉植物に保存され、環境鉄の変化に反応する。これらの翻訳抑制性uORFは、作物の鉄栄養および/または生物的ストレス耐性を改善することを目的とした新しい育種のための有用なターゲットである。
論議
Ribo-Seq データセットは、ペプチドおよび非ペプチド保存 uORF を予測するのに有用である。
uORF を同定するための相同性ベースの手法(uORFinder、uORFSCAN、BAIUCAS/ESUCA)がいくつか存在する。しかし、進化が新しく、配列保存性が低いuORFや非保存ペプチドuORF(uORFの最大のグループ)は、種を超えて保存された非共通アミノ酸配列を分析しても検出されない。暗号化された翻訳イベントの検出を可能にするリボソームロファイリングの開発は、uORFの同定に特に有用である。しかし、特にuORFは非正規の開始コドンを含む可能性があり、どのフレームにも存在する可能性があるため、ribo-seqデータセットからuORFの正確な開始部位を予測することは困難である。伸長するリボソームの3ヌクレオチド周期性を利用すれば、理論的には80Sリボソームの正確なPサイトを決定することができ、翻訳フレームと翻訳ペプチドを決定することができるが、1ヌクレオチド分解能のリボ-seqデータセットは、多くの実験的理由から作成が困難である。その代わりに、リボソームプロファイリングを使ってuORFのおおよその位置を推定し、その後、非正規開始点(すなわち停止コドンから停止コドン)を持つ可能性のあるすべてのORFの配列と保存解析を行った。これとは対照的に、psORFプラットフォームは、ノイズや低品質データのためと思われる低い成功率で、複数のribo-seqデータセットを用いてuORFの正確な開始部位を予測する(図2)。
例えば、psORFが予測したOsIDEF1の翻訳開始部位(CTG)は5'LSの中央に存在したが、OsIDEF1のリボソーム・プロファイルは翻訳が5'末端に向かって起こっていることを示していた(図1C)。さらなる配列解析の結果、LSの5'末端に2つ、3'末端に2つのカノニカル開始点(ATG)が同定され、5'末端の2番目のATGがmORFの大幅な抑制に関与し、他のATGはほとんど冗長であることがわかった(図4)。第2のATGは9bpの読み取り枠を持ち、5'キャップの7bp下流とmORF開始部位の265bp上流から始まる。リボソームプロファイル(図1D)と保存解析(図5A)を裏付けるように、psORFプラットフォームは、リボソームプロファイリングで見つかったのと同じATG uORF開始部位をOsIDEF2の5'LS内に同定した。対照的に、CTG開始部位(図1D)は単子葉植物間の配列保存性が低いため、真のuORF開始部位である可能性は低い(図5A)。したがって、uORFの同定を目的とした今後の研究プロジェクトでは、機能的DLAテストと並行してpsORFプラットフォームを活用し、その存在、位置、開始部位を決定すべきである。IDEF2-uORF領域に異なる参照枠を持つuORFが存在する可能性は排除できないが、翻訳抑制的な主要uORFは、より好ましい配列コンテキスト(すなわちATG開始部位)を含む可能性が高い(図5A)分析したすべての単子葉植物において、長さ90bpで29個の高度に保存されたアミノ酸を持つATG開始IDEF2-uORFが検出された(図5A)。一般に、このuORFは、ほとんどの単子葉植物IDEF2の5'LSに存在する第2エクソンの5'末端から20bp下流、mORF開始部位から約529bp上流に現れる。興味深いことに、OsIDEF2リボソームプロファイル(図1D)では、この〜529bpの領域内に2つの小さなピークが存在し、ATG開始uORF(92.6%ペアワイズ同一性)に対して単子葉植物間で保存性が低い(74.3%ペアワイズ同一性)。TaIDEF2 WT E1(〜529配列なし)とTaIDEF2 m4(〜529配列あり)を比較したDLAは同レベルの脱落を示し、2つの小さな余分なリボソームピークはノイズか、OsIDEF2 5'LSにのみ存在する弱い非保存uORFであることが示唆された(図5B)。同様に、本研究で解析した他の7つのFeホメオスタシス遺伝子の5'LS領域にも中程度のリボソームピークが検出された。これらの遺伝子におけるuORFの存在と機能を確認するには、最終的にDLA(Dynamic Load Assessment Test、DLAテスト)実験が必要となる。
(翻訳者注:Ribo-Seq(リボソームプロファイリングまたはリボソームシーケンシング)は、リボソームが翻訳中のmRNA断片を解析する技術です。この手法により、細胞内で実際に翻訳されている遺伝子やその翻訳量を網羅的に捉えることができます。Ribo-Seqは主に以下のような特徴と用途があります。
1. リボソーム結合mRNAの解析:細胞内でリボソームが結合しているmRNA断片のみを取得することで、実際に翻訳中のRNAを高解像度で特定できます。これは、通常のRNA-Seqでは把握しにくい「翻訳中」のmRNAの量や位置を可視化する点で重要です。
2. 翻訳開始部位の特定:翻訳が始まる場所(開始コドン)や、翻訳の終わり(終止コドン)の正確な位置を特定でき、代替的な翻訳開始点や小さなオープンリーディングフレーム(sORFs)も検出可能です。
3. タンパク質生成量の推定:mRNA上のリボソームの密度から、各mRNAがどの程度の頻度で翻訳されているか(翻訳効率)を推測し、実際のタンパク質生成量の見積もりに役立てます。
4. 翻訳調節の解析:ストレス条件下や細胞周期における翻訳の変動をモニタリングし、細胞が異なる条件下でどのようにタンパク質合成を調節しているかを理解します。
Ribo-Seqは、ゲノム全体の翻訳状態を高解像度で解析できるため、遺伝子発現調節の新しい層を理解するための強力なツールであり、基礎生物学や医療分野でも活用されています。)
uORF修飾による遺伝子発現制御は、将来の植物育種に応用できる可能性がある。
世界人口は2050年までに97億人に達すると予想されており、世界の作物生産は、増大する食糧需要を満たすために35〜56%増加する必要がある。部位特異的ヌクレアーゼ1(非相同末端結合)CRISPR-Cas9法は、遺伝子ノックアウトには効果的であるが、遺伝子発現の正確な増減はより困難である。上流のORFは、標的遺伝子の発現を微調整するために利用することができる。例えば、塩基編集とプライム編集を利用して、イネにde novo uORFをノックインすることで、遺伝子翻訳を高度に予測可能な形で減少させることができる。私たちのTaIDEF2-uORF変異シリーズ(図5)は、既存のuORFを段階的に変異させることにより、遺伝子翻訳を段階的に増加させる能力をサポートし、IDEF遺伝子におけるuORFの同定は、私たちのuORF同定ワークフローの有用性と実用性を示している。また、IDEF遺伝子におけるuORFの同定は、我々のuORF同定ワークフローの有用性と実用性を示している。
以下すべての図の全訳をしておきます。
図1
栄養恒常性に関与するイネ転写産物のリボソーム・プロファイル。(A)OsFER2、(B)OsGGP、(C)OsIDEF1、(D)OsIDEF2転写産物は、5'LS内のリボソームカバレッジにばらつきがあり、非正規翻訳の可能性に対応している。翻訳フレーム1(緑)、2(青)、3(紫)内の正規のATG開始コドンは、x軸に沿ってドットで表されている。転写産物モデルはリボソームプロファイルの下に、5'LS(左側の水平線)、コード配列(色のついたボックス)、3'UTR(右側の水平線)、および5'LS内に示された推定uORF(オレンジ色のボックス)で表されている。
翻訳者コメント1:「リボソームカバレッジ」という表現は、mRNAのどの部分がリボソームによって「カバーされている」か、つまりリボソームが結合して翻訳中である領域を指します。具体的には、リボソームが翻訳しているmRNAの範囲や密度を解析することで、どの遺伝子がどの程度翻訳されているのか、また、翻訳の開始位置や終了位置がどこかといった情報を取得できます。
翻訳者コメント2:アップストリームオープンリーディングフレーム(uORF)は、遺伝子の5'リーダー配列(5’LS)内のシス制御要素であり、一般にメインオープンリーディングフレーム(mORF)の翻訳を抑制する。
図2
イネの様々な鉄ホメオスタシス5'LSのデュアルルシフェラーゼアッセイ。(A) ホタルルシフェラーゼコード配列の上流と35Sプロモーターの下流に融合したWTまたは変異型(m1/m2)5'LSの模式図(左パネル)。推定される正規および非正規の開始コドンを青線で示し、5'LS内の停止コドン(赤線)を示した。ボックスプロット(右パネル)は、WT 5'LS(濃い緑)とその変異バージョン(薄い緑)の輝度比(ホタル/レニラ)(:firefly/renilla)を比較したものである。p値は、不等分散を仮定した2標本Students t-testによって決定された、同じ遺伝子の変異型とWT LSを比較したものである(n=5)。各生物学的連数は、1枚の葉の3つの浸潤部位から得られた3枚のリーフディスク(平均)で構成された。光度比(ホタル/レニラ)(:firefly/renilla)は、35 Sベクター(5'LSを含まない陽性対照)に対して正規化した。空ベクターは、プロモーターを含まないホタルルシフェラーゼコード配列を含む。35S浸潤(左上)、空の浸潤(右上)、および(B)OsIDEF1 WT(左下)およびOsIDEF1 m2(右下)、または(C)OsIDEF2 m1(左下)およびOsIDEF2 m1(右下)を含む、独立して浸潤されたN. benthamianaの葉の視覚的表現。
図3
イネ由来5'LSのデュアルルシフェラーゼアッセイ。ホタルルシフェラーゼコード配列の上流と35Sプロモーターの下流に融合したWTまたは変異(m1/m2)5'LSの模式図(左パネル)。推定される正規および非正規の開始コドンを青線で示し、5'LS内の停止コドン(赤線)を示した。ボックスプロット(右パネル)は、コントロールの単独浸潤(水色)またはFeSO4との共浸潤(紺色)のいずれかとして、様々な5'LSの光度比(ホタル/レニラ)(:firefly/renilla)を比較し、Feホメオスタシスを擾乱した。p値は、不等分散を仮定した2標本Students t-testによって決定された、コントロールと共浸潤5'LSを比較したものである(n=4)。各生物学的連数は、1枚の葉の3つの浸潤部位からの3枚の葉ディスク(平均)で構成された。光度比(ホタル/レニラ)(:firefly/renilla)は、35 Sベクターに対して正規化せず、FeSO4共浸潤がFe関連5'LSの添加なしにルシフェラーゼ遺伝子と相互作用しないことを示した。
図4
イネ由来OsIDEF1 5'LSのデュアルルシフェラーゼアッセイによるuORF開始部位の同定。ホタルルシフェラーゼコード配列の上流と35 Sプロモーターの下流に融合した変異ATGの組み合わせを含むOsIDEF1 5' LSの模式図(左パネル)。推定される正規の開始コドンを青線で示し、5'LS内の停止コドン(赤線)を示した。ボックスプロット(右パネル)は、様々な5'LSの輝度比(ホタル/レニラ)(:firefly/renilla)を比較したものである。文字は、一元配置分散分析(ポストホックTukey's HSD検定、n=5)により決定された、すべてのLS(コントロールを除く)間の有意差(p<0.05)を示す。各生物学的連数は、1枚の葉の3つの浸潤部位からの3枚の葉ディスクで構成された。光度比(ホタル/レニラ)(:firefly/renilla)は、35 Sベクター(5'LSを含まないポジティブコントロール)に対して正規化した。
図5
パンコムギ由来TaIDEF2 5'LSのデュアルルシフェラーゼアッセイ。 (A)単子葉植物におけるIDEF2オーソログ配列間のuORF含有領域のヌクレオチドアラインメント。E2の5'末端内に位置するuORF含有領域は、図1Dのリボソームピークと、5'LS内の保存されたORF(停止から停止まで)の解析に基づいて同定された。保存された正規の開始コドン(黄色の矢印)および保存された停止コドン(赤色の矢印)が、uORFの開始/停止部位として示されている。赤い四角はDLAに使用された5'LSを示す。(B)ホタルルシフェラーゼコード配列の上流および35Sプロモーターの下流に融合したWTまたは変異5'LSの模式図(左パネル)。TaIDEF2 5'LSは2つのエクソン(E1とE2)からなる。uORFを含む領域が示されている(オレンジ色の枠)。ボックスプロット(右パネル)は、様々な5'LSの輝度比(ホタル/レニラ)(:firefly/renilla)を比較している。文字は、一元配置分散分析(ポストホックTukey's HSD検定、n=4)により決定されたすべてのLS(コントロールを除く)間の有意差(p<0.05)を示す。光度比(ホタル/レニラ)(:firefly/renilla)は、35 Sベクター(5'LSを含まない陽性対照)に対して正規化した。(C)35S浸潤(左上)、空のベクター浸潤(右上)、TaIDEF2 WT(左下)、およびTaIDEF2 m3(右下)を含む、独立して浸潤されたN. benthamianaの葉の視覚的表示。
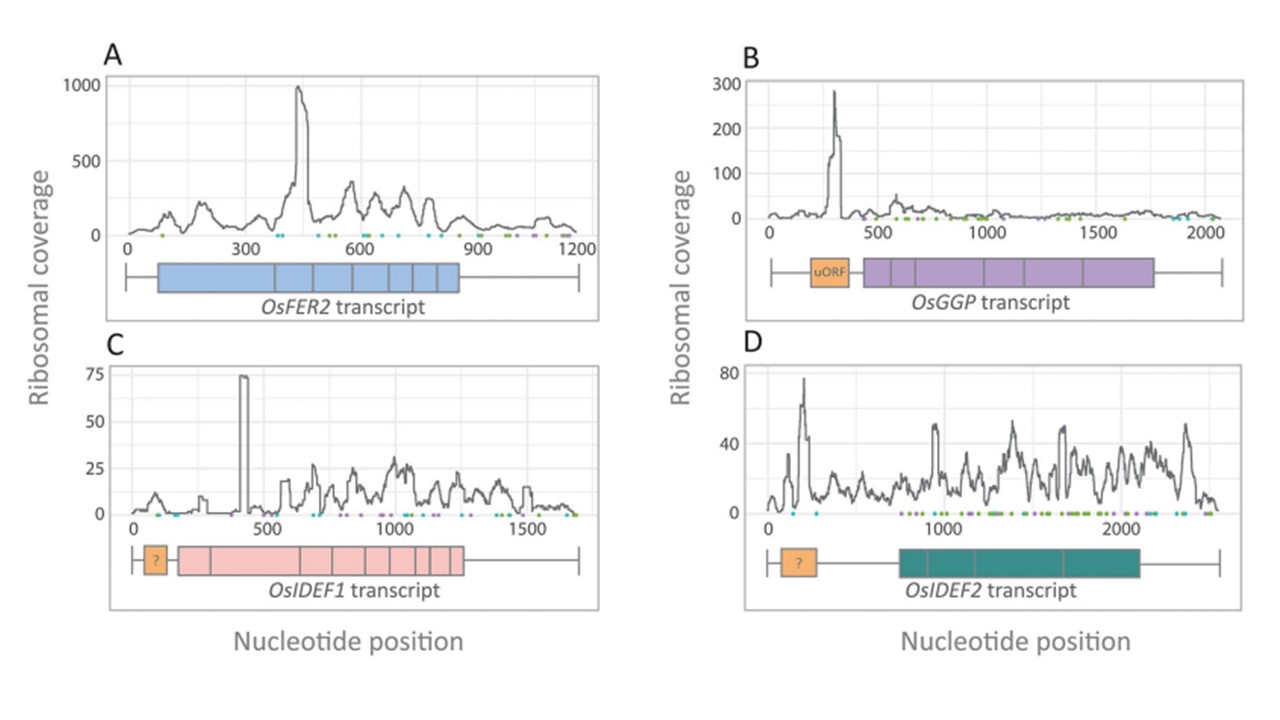
図1
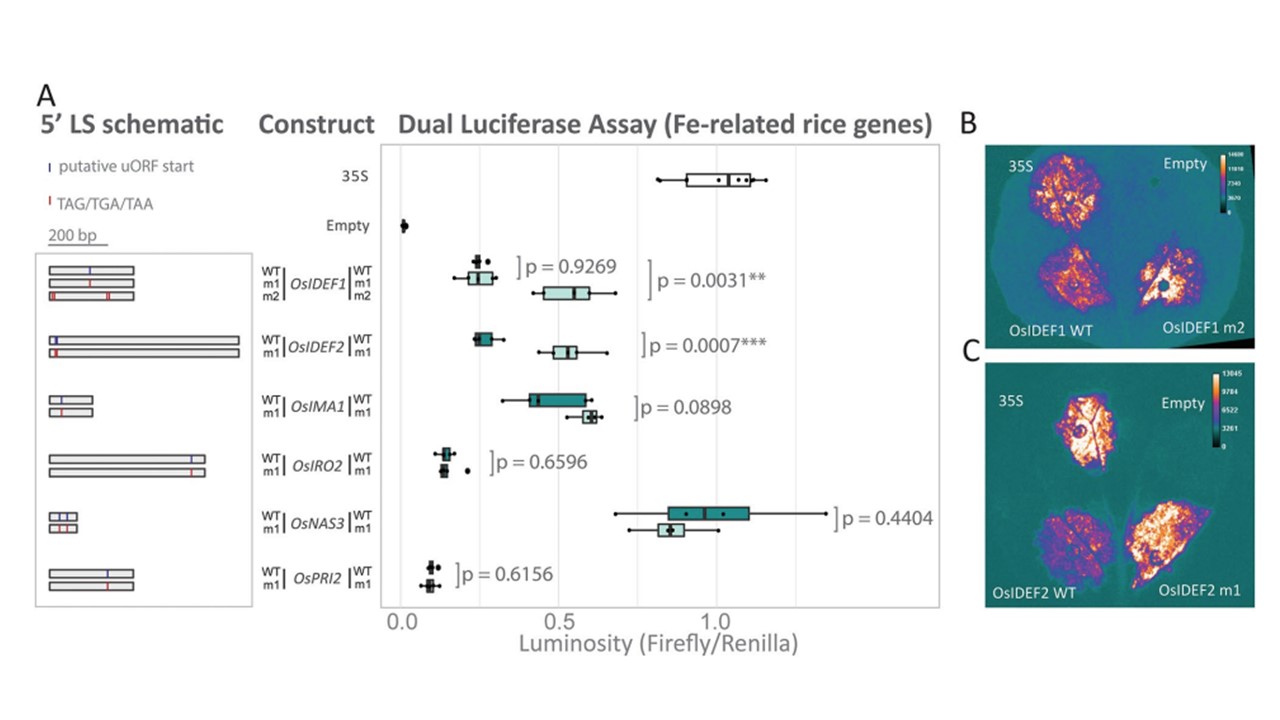
図2
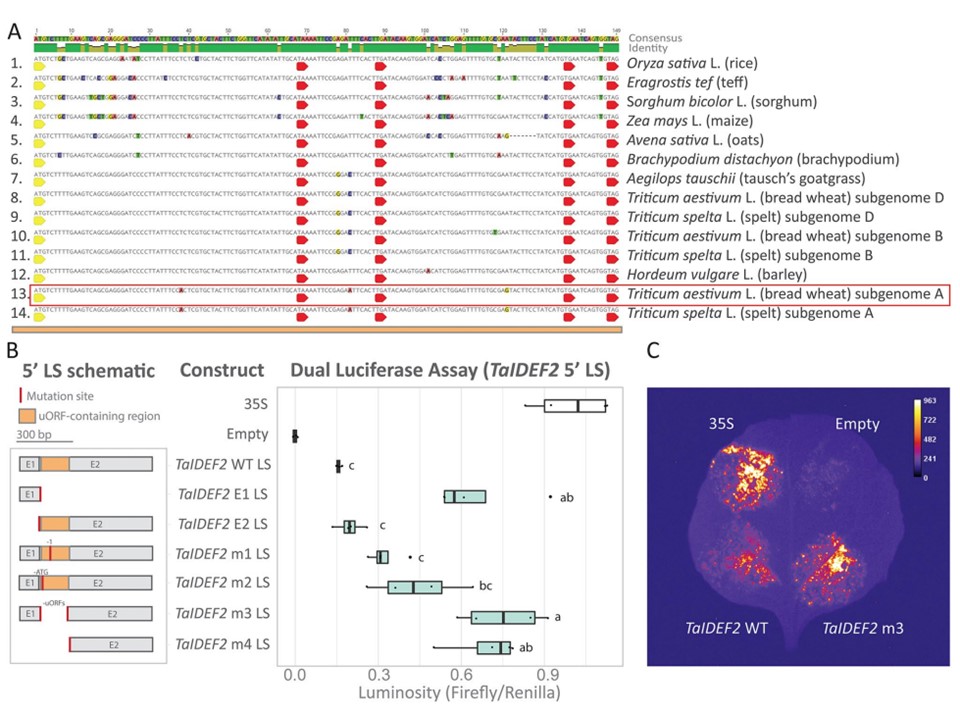
図5